タンパク質間相互作用 (protein-protein interaction; PPI) のデータベースであるSTRING (Search Tool for the Retrieval of Interacting Genes/Proteins)。以下は、そのデータを使ってRでネットワーク解析してみた個人的な備忘録です。大部分は"STRINGdb Package Vignette (15 March 2015)"に依拠してます。
まずSTRINGdbをインストールする
BioconductorからSTRINGdbをインストール。
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("STRINGdb", version = "3.8")
これはver 3.5以上のRの場合。それ以前の場合は知りません。
まず特定の生物種のPPI network情報をゲット
PPI network情報をロードします。ここではzebrafish (Danio rerio; taxid = 7955) を対象にします。
library(STRINGdb)
string_db <- STRINGdb$new (version="10", species=7955,
score_threshold=0, input_directory="" )
コマンド中のversion=10はSTRINGのversionを示し、7955はzebrafishのtaxidを示しています。score_thresholdは、ロードするタンパク質相互作用のスコア下限です。STRINGではタンパク質間の関係をcombined scoreなる指標で表現しているそうですが(参考:FAQ)、ここでは指定したcombined score未満のPPIはロードしません。デフォルト値は400だそうです。
次にネットワークとして表現します。下のコマンドでグラフ形式に変換?できます。
full.graph <- string_db$get_graph()
full.graphの中身を見ると、こんな感じ。23,369の頂点verticesと9,145,627の辺edgesが含まれています。
> full.graph
IGRAPH d850ca6 UN-- 23369 9145627 --
+ attr: name (v/c), neighborhood (e/n), neighborhood_transferred
| (e/n), fusion (e/n), cooccurence (e/n), homology (e/n),
| coexpression (e/n), coexpression_transferred (e/n), experiments
| (e/n), experiments_transferred (e/n), database (e/n),
| database_transferred (e/n), textmining (e/n),
| textmining_transferred (e/n), combined_score (e/n)
+ edges from d850ca6 (vertex names):
[1] 7955.ENSDARP00000000004--7955.ENSDARP00000051551
[2] 7955.ENSDARP00000000192--7955.ENSDARP00000023805
[3] 7955.ENSDARP00000000192--7955.ENSDARP00000023951
+ ... omitted several edges
attrはfull.graphの属性のことで、()内にvとあるのが頂点に関するもの、eとあるのが辺に関する属性です。()内にcとあるのが文字characterで表現された属性、nは数値numericで表された属性。例えばnameは文字で表現された頂点に関する属性。
属性は下のようなコマンドで取り出せます。
V(full.graph)$name
igraphパッケージで描画
全部を描くと大変なので、次数の大きい頂点トップ50個を選びます。
top.vertices <- names( tail(sort(degree(full.graph)), 50) )
top.subgraph <- induced_subgraph(full.graph, top.vertices)
plot(top.subgraph,vertex.label=NA,vertex.size=10)

同じネットワーク図を、STRING独自のスタイルで描画することもできます。
string_db$plot_network(V(top.subgraph)$name)
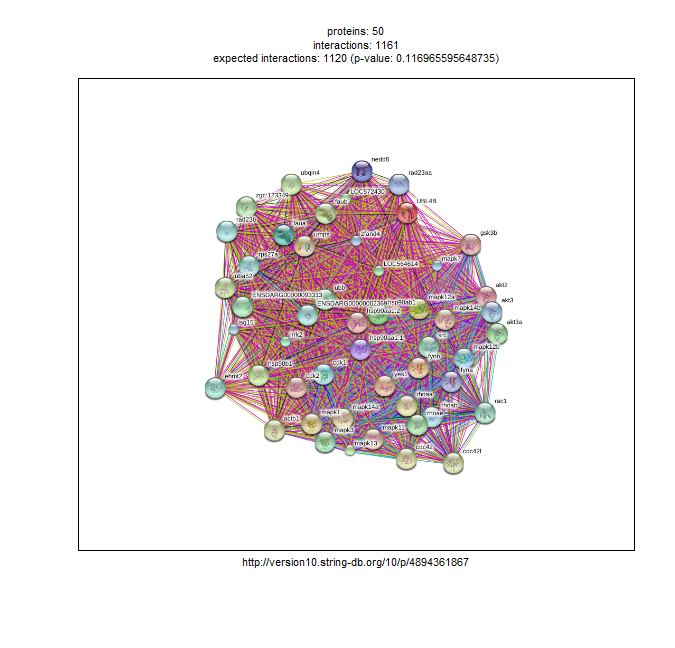
STRING形式の図は、カラフルで色んな情報が含まれてます。これはブラウザ上でSTRINGを利用した際にも見ることが出来ます。
ただ少し複雑すぎるので、注目したい情報だけ取り出して描画してみます。
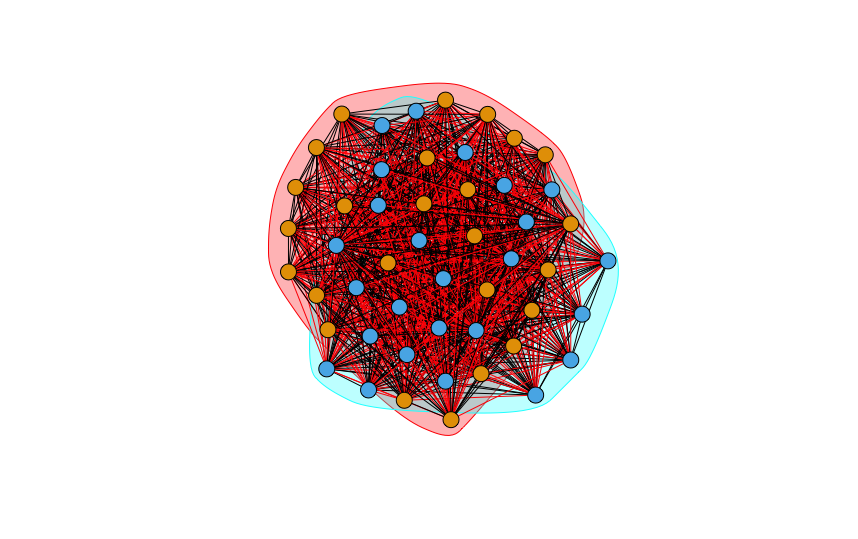
例えば、STRINGの相互作用情報にはcooccurenceがあります。ネットワークの辺の太さをcooccurenceの程度によって変えてみます。※単なる描画の練習で、生物学的な意味は考慮していません。
E(top.subgraph)$width <- log10( E(top.subgraph)$cooccurence )
plot(top.subgraph,vertex.label=NA,vertex.size=10)

同じ要領でV(top.subgraph)$colorによって色を変えたり、E(top.subgraph)$ltyで辺の実線・点線を変えたりできます。
STRINGdbパッケージでは、get_annotations()でGO (Gene Ontology) やKEGG pathwayなどタンパク質のアノテーションを得ることもできます。
Annotation <- string_db$get_annotations()
> head(Annotation)
STRING_id term_id category type
1 7955.ENSDARP00000000004 GO:0006810 Process IEA
2 7955.ENSDARP00000000004 GO:0006811 Process IEA
3 7955.ENSDARP00000000004 GO:0006812 Process IEA
4 7955.ENSDARP00000000004 GO:0006818 Process IEA
5 7955.ENSDARP00000000004 GO:0008150 Process IEA
6 7955.ENSDARP00000000004 GO:0008643 Process IEA
また、enrichment解析もできるそうです。例えば↓。
test<-string_db$get_enrichment(top.verticies)
> head(test)
term_id proteins hits pvalue pvalue_fdr term_description
1 GO:0044267 434 10 1.542945e-17 6.403222e-15 cellular protein metabolic process
2 GO:0019538 478 10 4.085650e-17 8.477723e-15 protein metabolic process
3 GO:0006468 71 7 2.615610e-16 3.618260e-14 protein phosphorylation
4 GO:0006950 190 8 1.080273e-15 1.120783e-13 response to stress
5 GO:0016310 116 7 9.115423e-15 7.565801e-13 phosphorylation
6 GO:0038083 4 4 1.690731e-14 1.169423e-12 peptidyl-tyrosine autophosphorylatio
STRINGdbパッケージでは、get_clusters()でクラスタリング解析もできます。
clustersList <- string_db$get_clusters( V(top.subgraph)$name, algorithm="fastgreedy")
クラスタリングのアルゴリズムは、fastgreedy、walktrap、spinglass、edge.betweennessの4種類から選べます。
> clustersList
1
[1] "7955.ENSDARP00000009659" "7955.ENSDARP00000011298" "7955.ENSDARP00000019813"
[4] "7955.ENSDARP00000020408" "7955.ENSDARP00000021095" "7955.ENSDARP00000034350"
[7] "7955.ENSDARP00000035686" "7955.ENSDARP00000037198" "7955.ENSDARP00000040361"
[10] "7955.ENSDARP00000060744" "7955.ENSDARP00000064110" "7955.ENSDARP00000069405"
[13] "7955.ENSDARP00000075784" "7955.ENSDARP00000076407" "7955.ENSDARP00000087028"
[16] "7955.ENSDARP00000087454" "7955.ENSDARP00000089879" "7955.ENSDARP00000093618"
[19] "7955.ENSDARP00000107110" "7955.ENSDARP00000107491" "7955.ENSDARP00000107646"
[22] "7955.ENSDARP00000111940" "7955.ENSDARP00000112153" "7955.ENSDARP00000123746"
[25] "7955.ENSDARP00000123888"
2
[1] "7955.ENSDARP00000009190" "7955.ENSDARP00000013441" "7955.ENSDARP00000014978"
[4] "7955.ENSDARP00000018692" "7955.ENSDARP00000022302" "7955.ENSDARP00000023119"
[7] "7955.ENSDARP00000026065" "7955.ENSDARP00000027785" "7955.ENSDARP00000032448"
[10] "7955.ENSDARP00000035728" "7955.ENSDARP00000038550" "7955.ENSDARP00000040644"
[13] "7955.ENSDARP00000053431" "7955.ENSDARP00000054986" "7955.ENSDARP00000058736"
[16] "7955.ENSDARP00000084288" "7955.ENSDARP00000084961" "7955.ENSDARP00000088354"
[19] "7955.ENSDARP00000094521" "7955.ENSDARP00000096950" "7955.ENSDARP00000103480"
[22] "7955.ENSDARP00000109027" "7955.ENSDARP00000109720" "7955.ENSDARP00000118902"
[25] "7955.ENSDARP00000119429"
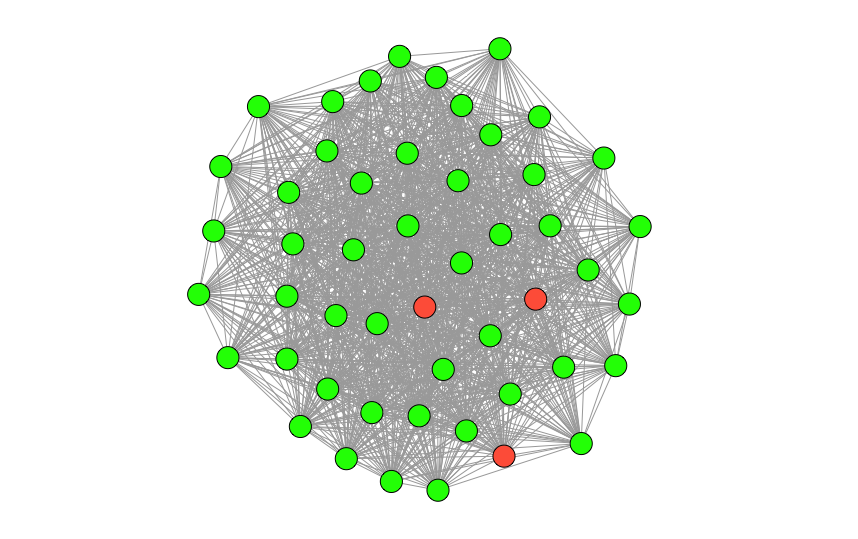
Cluster 1に含まれる頂点25個を緑、Cluster 2のものをトマト色で描画します。
V(top.subgraph)$ color <- ifelse ( is.na(match(V(top.subgraph)$name, clustersList1)), "green", "tomato" )
plot(top.subgraph,vertex.label=NA,vertex.size=10)
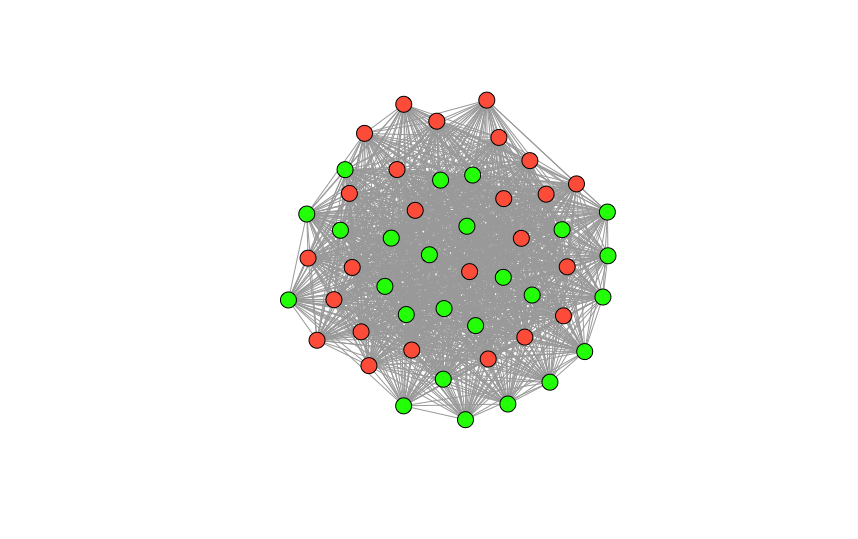
上記はSTRINGdb内の関数を使用しましたが、igraphパッケージの関数を使用して同様のクラスタリングをおこなうことも可能です。
clusters <- cluster_fast_greedy(top.subgraph)
plot(clusters,top.subgraph,vertex.label=NA,vertex.size=10)