前回の続き。今回は、STRINGdbから得たPPI networkの情報を、DEG解析(Differentially Expressed Genes)の結果と絡める方法について書きます。
前回までの話
ゼブラフィッシュのPPI networkをSTRINGdbから取得します。例えばこんな感じのデータ。
> full.graph
IGRAPH a1f4259 UN-- 23369 9145627 --
+ attr: name (v/c), neighborhood (e/n), neighborhood_transferred (e/n), fusion (e/n),
| cooccurence (e/n), homology (e/n), coexpression (e/n), coexpression_transferred (e/n),
| experiments (e/n), experiments_transferred (e/n), database (e/n), database_transferred
| (e/n), textmining (e/n), textmining_transferred (e/n), combined_score (e/n)
+ edges from a1f4259 (vertex names):
[1] 7955.ENSDARP00000000004--7955.ENSDARP00000051551 7955.ENSDARP00000000192--7955.ENSDARP00000023805
[3] 7955.ENSDARP00000000192--7955.ENSDARP00000023951 7955.ENSDARP00000000192--7955.ENSDARP00000024039
[5] 7955.ENSDARP00000000192--7955.ENSDARP00000024056 7955.ENSDARP00000000192--7955.ENSDARP00000024217
[7] 7955.ENSDARP00000000192--7955.ENSDARP00000024581 7955.ENSDARP00000000192--7955.ENSDARP00000024929
[9] 7955.ENSDARP00000000192--7955.ENSDARP00000025540 7955.ENSDARP00000000192--7955.ENSDARP00000026031
+ ... omitted several edges
タンパク質名の変換
STRINGでは上記の通り、"ENSDARP00000000004"のようなEnsembleのタンパク質名で表現されています。けれどDEG解析をした場合など、手元にあるのは遺伝子名や転写産物名のリストであることも多いでしょう。その場合、名称を変更する必要があります。
STRINGにはalias情報が入っているので、容易に変更できます。
まずalias情報を取りだします。
Ali <- string_db$get_aliases()
> head(Ali)
STRING_id alias
1 7955.ENSDARP00000003766 plek
2 7955.ENSDARP00000035258 tesca
3 7955.ENSDARP00000045815 plg
4 7955.ENSDARP00000076522 gsc
5 7955.ENSDARP00000117101 aftpha
6 7955.ENSDARP00000126089 SEPT12
DEG解析の結果、下の6つの遺伝子が得られ、これらの遺伝子がPPI netowkrのどこに位置するのかを調べたいとします。
Gene <- c("pik3r3a","arap1a","UBL4B","yes1","hsp90ab1","zgc:158404")
matchコマンドを使って、Geneとaliasなタンパク質を探します。
> Gene2 <-Ali[match(Gene,Ali$alias),]
> Gene2
STRING_id alias
17386 7955.ENSDARP00000106632 pik3r3a
NA <NA> <NA>
833 7955.ENSDARP00000009190 UBL4B
879 7955.ENSDARP00000009659 yes1
1461 7955.ENSDARP00000014978 hsp90ab1
12133 7955.ENSDARP00000093251 zgc:158404
大方の遺伝子はaliasが見つかってますが、2つ目は見つかっていません。aliasが見つからない場合は、遺伝子名ではなく例えばensembleのid(ENSDARG....)に変換してから探すと良いかも。
(2021.01追記) 例えばRならbiomaRtパッケージのgetBMでensembl gene idからensembl protein idに変換できます。
ネットワークの描画
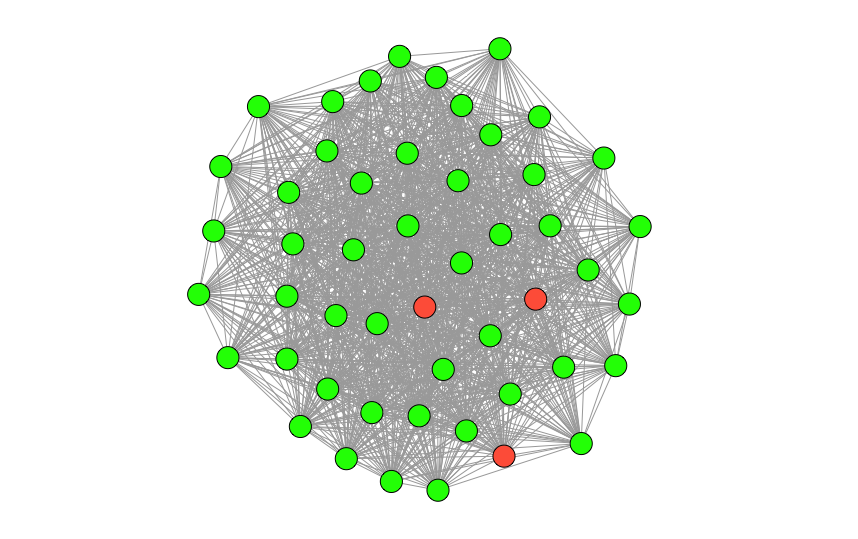
V(full.graph)$ color <- ifelse ( is.na(match(V(full.graph)$name, Gene2$STRING_id)), "green", "tomato" )
Geneの6遺伝子とaliasであるタンパク質はトマト色、それ以外の遺伝子は緑色で示すことにします。
そして前回と同様にigraphパッケージで描画します。
top.vertices <- names( tail(sort(degree(full.graph)), 50) )
top.subgraph <- induced_subgraph(full.graph, top.vertices)
plot(top.subgraph,vertex.label=NA,vertex.size=10)
"Gene"リストに含まれていたタンパク質は赤色で表示されています。
このあとは、例えばenrichment解析などに進みます。