重み付き遺伝子共発現ネットワーク解析(WGCNA; weighted gene co-expression network analysis)のはなし。
「遺伝子共発現ネットワークによるDaphniaの繁殖影響予測」
Asselman J, Pfrender ME, Lopez JA, Shaw JR, De Schamphelaere KAC, 2017, Gene co-expression networks drive and predict reproductive effects in Daphnia in response to environmental disturbances, Environmental Sci Technol, in press.
殺虫剤とシアノバクテリアのbinary mixtures (全144サンプル) に曝露させたミジンコのマイクロアレイデータと産仔数データを、WGCNAで統合して分析してます。目的は、遺伝子発現データからmixture exposureの産仔数を予測したい、というもの。産仔数と相関のない遺伝子モジュールを省いていき、予測精度を上げていく流れ。最終的には一般化加法モデル (GAM) で予測。
Mixureだと産仔数の予測精度が低下するなど結果自体の新鮮さはあまりないかもですが、モデルの交互作用項で遺伝子発現レベルの複合影響を考慮するという手法など面白い。
「オオミジンコの初期転写反応は甲殻類特異的な遺伝子ネットワークに位置する」
Orsini L, Brown JB, Shams Solari O, Li D, He S, Podicheti R, Stoiber MH, Spanier KI, Gilbert D, Jansen M, Rusch D, Pfrender ME, Colbourne JK, Frilander MJ, Kvist J, Decaestecker E, De Schamphelaere KAC, Meester LD, 2017, Early transcriptional response pathways in Daphnia magna are coordinated in networks of crustacean specific genes, Mol Ecol, in press.
オオミジンコDaphnia magnaを12種のストレス(シアノバクテリア, 金属, 殺虫剤, 低pHなど)にそれぞれ曝露させ、RNA-seqした論文。オオミジンコは3つのgentotypeを使用してます。
この論文の結果の一つは、2011年ScienceのD. pulexゲノム論文の確認。 すなわち、環境ストレスによって発現変動した遺伝子は甲殻類特異的だということ。さらにgenotypeにも特異的だそうです。この論文では甲殻類crustaceansと言っているけど実際はD. pulexとD. magnaしか相同性検索に使用してないので、環境ストレス応答遺伝子が系統によって異なるのはDaphniaに限った話じゃないかと思ったのですが、C. elegansでも報告されている話らしい(Zhou et al., 2015, Genomics)。
ストレス曝露によって発現変動した遺伝子の解析(DEG解析)では、キチン・表皮の考察話が多い。これは正直知見が多くて、議論しやすいから議論されてるだけな気もしますが。Zebrafishのメタ解析の論文と同じく、DEG解析ではあまり多くのストレス応答遺伝子を見つけられなかったので(全処理で変動したのは1,396genes; およそ全3万遺伝子中)、追加でWGCNA解析をしてます。WGCNAでモジュールを調べることで、アノテーションのない遺伝子の機能を推測できるかも、という話ですが、いまいち結論が見えにくい…。データが膨大過ぎて解析が追いついていない感。
以下、WGCNAの勉強。
超絶初心者の自分用メモです。ご容赦ください。
共起発現ネットワークについては、英語版wikiが分かりやすい。発現量の相関係数sijを計算し、その相関係数がある閾値を超えたら1、閾値以下なら0というルールに従って隣接行列に変換する。
ただWGCNAでは、隣接行列は2値(0 or 1)ではなく、連続値(相関係数sijの累乗sij^a)を用いるそうです。ここが"weighted"たる所以だそうな。どうやって指数aを求めるかは元論文のZhang and Horvath (2005) を見ましょう(適当)。RのWGCNAパッケージだとpickSoftThreshold関数で求められるっぽい。
モジュール(=関連のある遺伝子の集まり)の決め方は普通の階層的クラスタリングでもOK。課題は、どこでクラスターを切るか。
以下、RのWGCNAパッケージを使った解析の練習。下のパッケージ開発の論文とその詳しいチュートリアル(ここ)とを見ながらやってみました。
Langfelder P, Horvath S, 2008, WGCNA: an R package for weighted correlation network analysis, BMC Bioinformatics 9(1): 559.
チュートリアルのデータは複雑なので、まずもっと簡単なデータとして先ほどの遺伝子共起発現ネットワークのwikiページのものを使います。
data <- matrix( c(43.26, 166.6, 12.53, 28.77, 114.7, 119.1, 118.9, 3.76, 32.73, 17.46, 40.89, 41.87, 39.55, 191.92, 79.7, 80.57, 156.69, 2.48, 11.99, 56.11, 5.05, 136.65, 42.09, 236.56, 99.76, 114.59, 186.59, 136.78, 118.8, 21.41), nrow=10, ncol=3)
rownames(data)<- c("G1","G2","G3","G4","G5","G6","G7","G8","G9","G10")
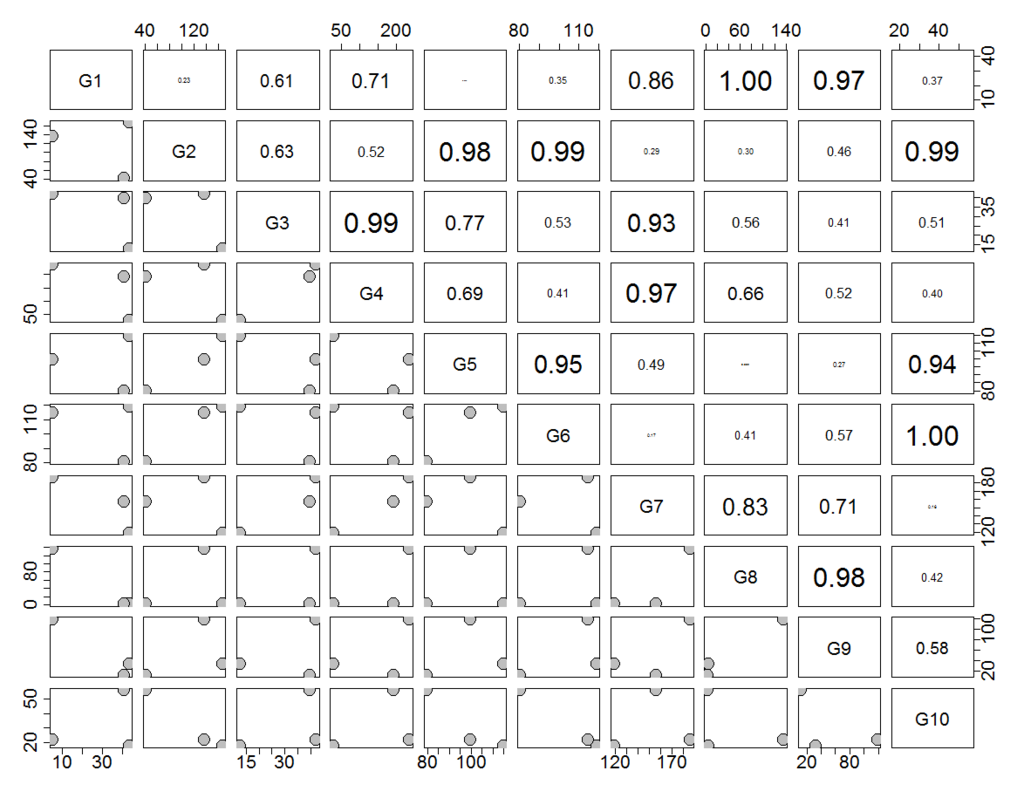
adj <- abs( cor(t(data) , method="pearson") )
隣接行列adjは次のようになる。
G1 G2 G3 G4 G5 G6 G7 G8 G9 G10
G1 1.000 0.234 0.610 0.706 0.029 0.353 0.860 0.998 0.971 0.366
G2 0.234 1.000 0.627 0.523 0.979 0.992 0.295 0.295 0.458 0.990
G3 0.610 0.627 1.000 0.992 0.774 0.525 0.929 0.559 0.405 0.513
G4 0.706 0.523 0.992 1.000 0.687 0.413 0.969 0.660 0.518 0.400
G5 0.029 0.979 0.774 0.687 1.000 0.945 0.485 0.092 0.265 0.941
G6 0.353 0.992 0.525 0.413 0.945 1.000 0.174 0.412 0.565 1.000
G7 0.860 0.295 0.929 0.969 0.485 0.174 1.000 0.826 0.714 0.160
G8 0.998 0.295 0.559 0.660 0.092 0.412 0.826 1.000 0.985 0.425
G9 0.971 0.458 0.405 0.518 0.265 0.565 0.714 0.985 1.000 0.577
G10 0.366 0.990 0.513 0.400 0.941 1.000 0.160 0.425 0.577 1.000
pairs(t(data),cex=3,cex.axis=2,pch=21,bg="grey")
始めは、重みづけなしの共起発現ネットワークで処理する。0.8を閾値とします。
adj2<- ifelse ( adj > 0.8, 1,0)
# sna packageで描画
library(sna)
gplot(adj2,usearrows=FALSE, displaylabels=TRUE)
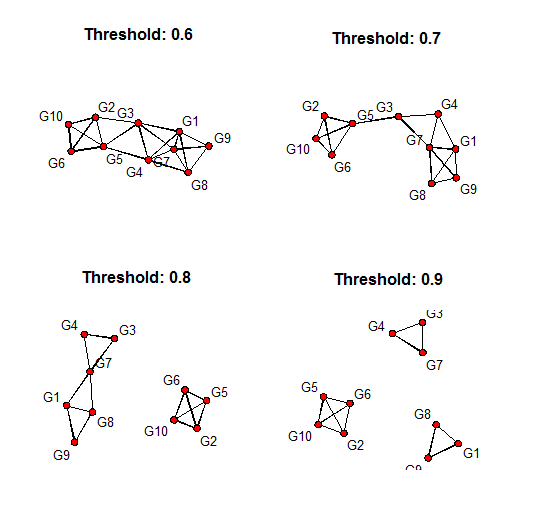
試しに閾値の取り方を変えてみると、ネットワークのつながり方も大きく変化します。
次にWGCNAパッケージを使用。
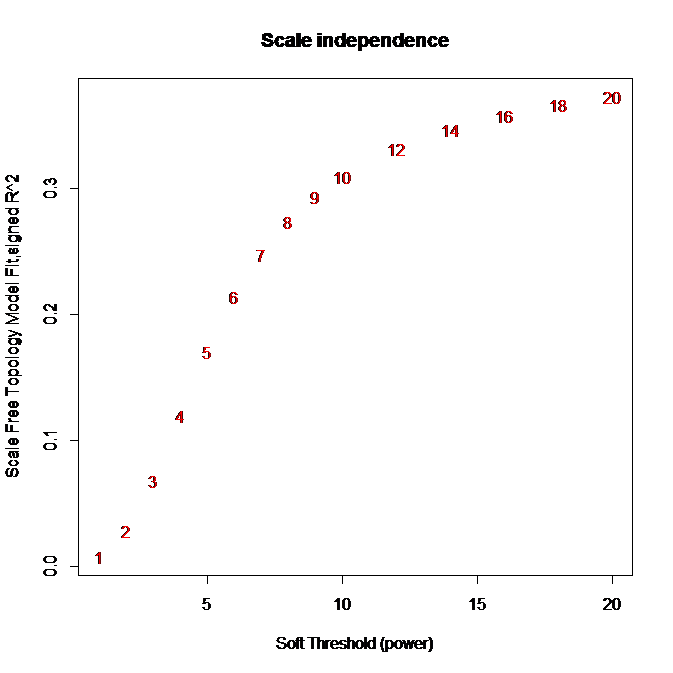
sft = pickSoftThreshold(data, dataIsExpr = TRUE)
# Sclale-free topology fit indexを描画
plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Soft Threshold (power)",ylab="Scale Free Topology Model Fit,signed R^2",type="n",
main = paste("Scale independence"))
text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
labels=powers,col="red")
20乗(power=20)してもScale-free topology fit indexが0.4程度と低いまま。FAQにその対処法が書かれてますが、今回は相関係数の絶対値を取っているunsigned networkなので、FAQに従いpowerを9とします。
このあたりから、かなり手探り状態…。
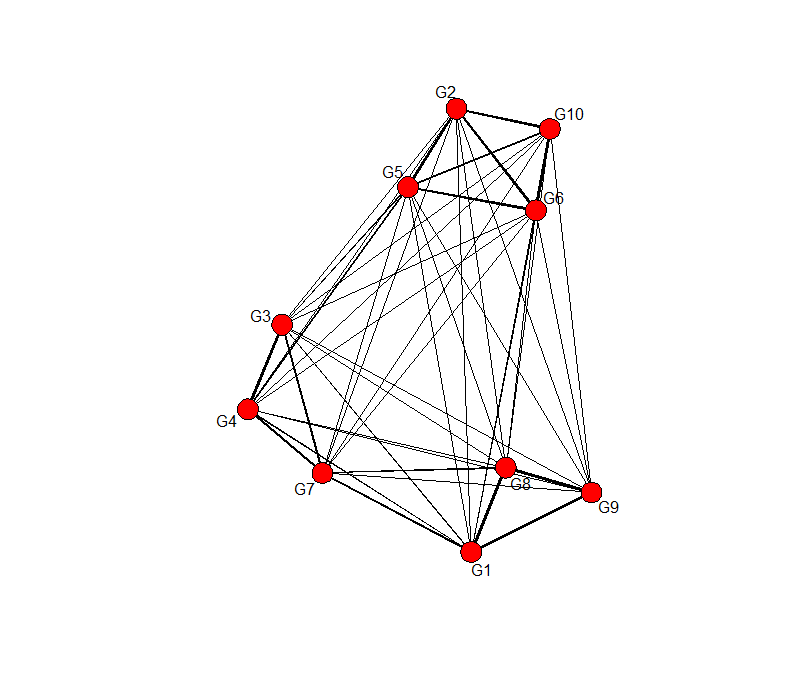
adj_w <- adjacency ( t(data),power=9 )
# snaで描画してみる, 辺の太さは隣接行列の大きさに従う
gplot(adj_w, usearrows=FALSE, displaylabels=TRUE, edge.lwd=adj_w*2)
太い辺edgeに注目したら、上のunweightedの場合のネットワークとほぼ同じですね。
次にクラスタリング。
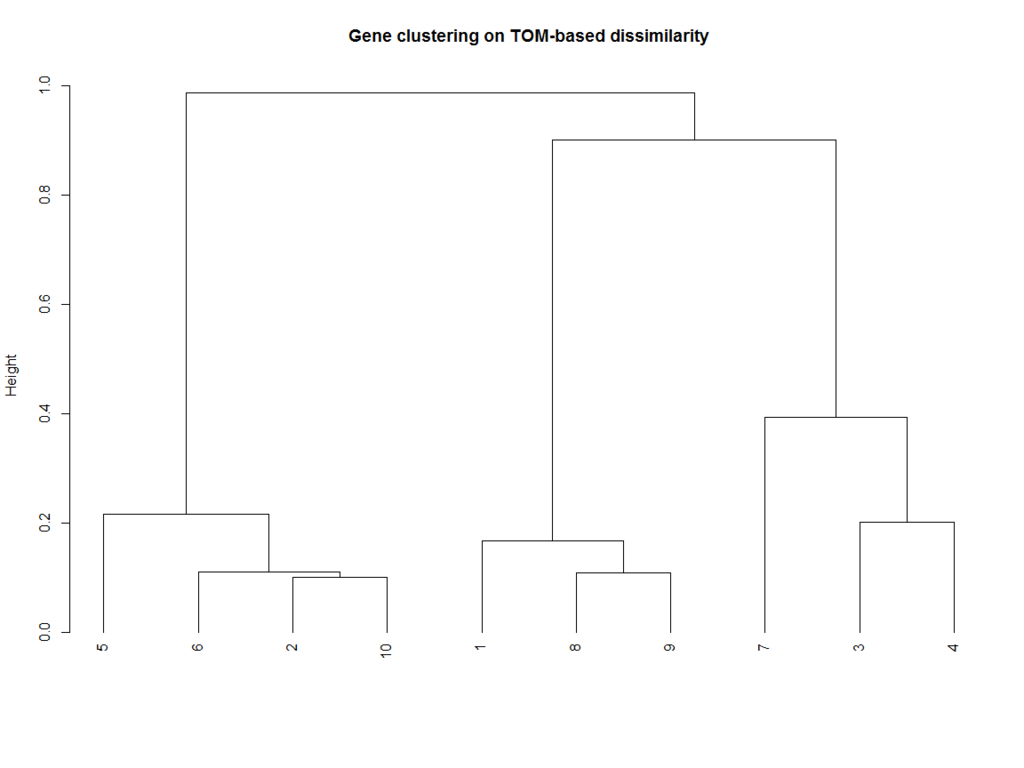
TOM = TOMsimilarity(adj_w)
dissTOM = 1-TOMgeneTree = hclust(as.dist(dissTOM), method = "average")
sizeGrWindow(12,9)
plot(geneTree, xlab="", sub="", main = "Gene clustering on TOM-based dissimilarity",hang =-1)
# 枝の剪定
dynamicMods = cutreeDynamic(dendro = geneTree, distM = dissTOM, deepSplit = 2, pamRespectsDendro = FALSE, minClusterSize=2)
dynamicColors = labels2colors(dynamicMods)dynamicColors
[1] "blue" "turquoise" "brown" "brown" "turquoise" "turquoise" "brown" "blue"
[9] "blue" "turquoise"
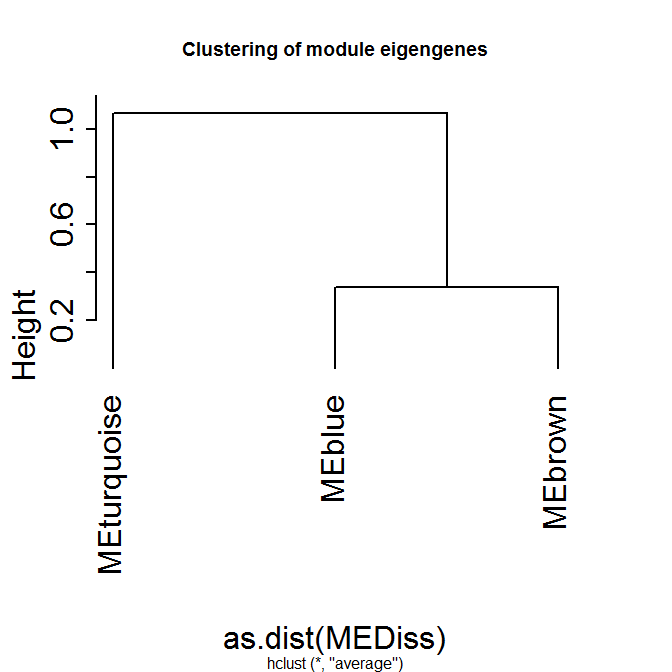
最後にeigengene。Eigengeneとは、各モジュールにおける第一主成分のことらしいです。モジュールを代表するような発現だと考えて良いみたい。
MEList = moduleEigengenes( t(data), colors = dynamicColors)
MEs = MEList$eigengenes
MEDiss = 1-cor(MEs)
METree = hclust(as.dist(MEDiss), method = "average")plot(METree, main = "Clustering of module eigengenes", hang=-1)
#blueはGene1,8,9で、brownはGene3,4,7。
疲れたので、とりあえずここまで。
あとは、遺伝子発現以外の情報(生態毒性だと致死率・繁殖阻害・成長阻害とか)と各モジュールとの関係を見るなどの解析をして、面白いモジュールを見つけるのが王道でしょうか。
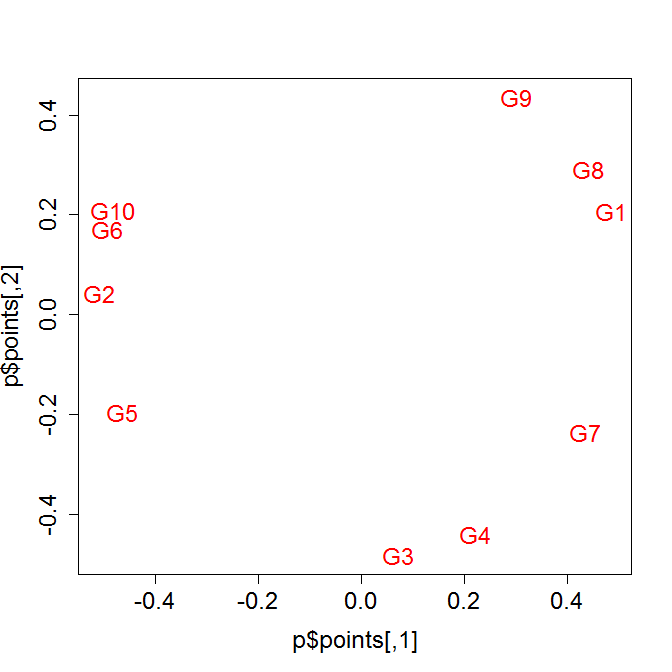
ちなみに同じ発現データの相関を取ってMDSプロットしてみた場合。
library(MASS)
p<- isoMDS( 1-abs(cor(t(data))), k=2, maxit=100)
plot(p$points, type="n",cex.axis=1.5,cex.lab=1.5)
text(p$points, rownames(p$points), col="red",cex=1.5)